领域前沿·中国
CBASS系统的研究进展
尹森 高昂*
(北京理工大学生命学院, 北京 100081)
 | 高昂, 北京理工大学生命学院教授、博士生导师。2005年本科毕业于山东大学;2008、2013年分别于山东大学微生物国家重点实验室和中国科学院生物物理研究所获硕士、博士学位; 2013年至2018年在美国纽约大学Langone医学院从事博士后研究; 2019年入选国家级青年人才计划。主要从事细菌与环境相互作用研究, 特别聚焦于环二核苷酸信使分子在细菌防御、代谢调控及与宿主互作中的关键核心功能。在相关领域以第一和通信作者(含共同)发表系列工作(Cell 2025, Nature 2022, Cell 2018, Nat Commun 2025, Nat Commun 2024, Mol Cell 2019, Nat Chem Bio 2014, Nucleic Acids Res 2020, Nucleic Acids Res 2018)。 |
CBASS系统的研究进展
尹森 高昂*
(北京理工大学生命学院, 北京 100081)
【摘要】亿万年的时间里, 噬菌体与细菌之间不断进行着攻击–防御–反击的战斗。在漫长的共同进化过程中, 噬菌体不断更新侵染系统来攻破细菌的防线。同时, 细菌为保护自己免受噬菌体的入侵, 进化出一系列复杂而精巧的抗噬菌体防御系统, 又被称为“原核生物的免疫系统”。CBASS系统作为原核生物重要的抗噬菌体防御机制, 其功能类似于真核生物中cGAS抗病毒的免疫系统,在原核生物中以古老又广泛存在的形式发挥着抗噬菌体作用。细菌受到噬菌体侵染后, CBASS系统中的寡核苷酸环化酶接收信号被激活生成环状寡合苷酸信号分子, 该信号分子可激活CBASS系统中的效应蛋白发挥其致死功能, 诱导宿主细胞死亡以阻断噬菌体扩散。该文介绍了原核生物中CBASS系统的发现, 各个组成部分及其功能和研究进展, 并展望了其在新型抗菌策略和生物技术工具开发中的应用前景。
【关键词】CBASS系统; 抗噬菌体防御; 环状寡核苷酸; 效应蛋白
Research Progress on the CBASS System
YIN Sen, GAO Ang*
(School of Life Sciences, Beijing Institute of Technology, Beijing 100081, China)
【Abstract】Over hundreds of millions of years, bacteriophages and bacteria have engaged in an ongoing“attack-defense-counterattack” battle. Through prolonged co-evolution, bacteriophages have continuously upgraded their infection systems to breach bacterial defenses. Meanwhile, bacteria have evolved a series of sophisticated and intricate anti-phage defense systems—also referred to as the “immune system of prokaryotes”—to protect themselves from phage invasion. As a crucial anti-phage defense mechanism in prokaryotes, the CBASS system functions analogously to the cGAS-mediated antiviral immune system in eukaryotes. It operates in an ancient yet widespread form to defend prokaryotes against phage attacks. Upon bacteriophage infection, the oligonucleotide cyclase in the CBASS system receives signals and becomes activated, generating cyclic oligonucleotide signaling molecules. These molecules then activate effector proteins within the CBASS system, triggering their lethal functions to induce host cell death and block phage dissemination. This review introduces the discovery of the CBASS system in prokaryotes, its components and functions, highlights recent research advances, and discusses its potential applications in developing novel antimicrobial strategies and biotechnological tools.
【Keywords】CBASS system; anti-phage defense; cyclic oligonucleotides; effector proteins
在细菌与噬菌体的长期进化博弈中, 噬菌体发展出了各种精准有效的攻击手段。而细菌也绝非坐以待毙的弱者, 它们同样进化出了多种多样的防御机制[1-2]。例如目前研究的较为透彻的限制–修饰(restriction-modification, R-M)系统和CRISPR-Cas系统[3-4]。限制–修饰系统可识别噬菌体基因组上特定DNA序列并对其进行切割, 同时又能对宿主自身DNA进行甲基化修饰使其免受降解[5-6]。CRISPRCas系统则是通过获取病毒DNA短片段并将其整合到宿主基因组中而获得免疫性记忆, 然后将这些序列转录并加工成为crRNA(CRISPR RNA), 并引导Cas核酸酶通过碱基互补识别并切割入侵的病毒核酸[7-8]。细菌防御系统的发现和对其分子机制的深入研究, 推动了重要的生物学工具的发展, 甚至整个生命科学领域革命性的进步。例如: 来源于细菌限制–修饰系统的限制性内切酶的发现, 引发了基因工程的革命, 其成为了分子克隆的必备工具; 而CRISPRCas技术已经彻底改变了基因编辑领域。
第二信使是细胞内信号转导的重要启动组成部件之一, 它们能够通过激活级联系统中酶等蛋白的活性, 控制细胞的生命活动。2019年, 以色列SOREK课题组[9]发现了一种新型的细菌防御系统—基于环化寡核苷酸的抗噬菌体信号系统(cyclic oligonucleotide-based antiphage signalling system,CBASS), 便是通过响应噬菌体感染产生环状寡核苷酸信号分子, 继而激活效应蛋白, 在噬菌体完成复制前诱导宿主细胞死亡这一“顿挫感染” 策略阻断其扩散[10]。这一系统不仅开拓了人们对原核生物免疫网络的认知, 更因其与真核生物cGAS-STING通路的进化同源性, 成为连接原核与真核免疫机制的关键纽带。本文将从CBASS系统的结构分类、作用机制、研究进展及应用前景等方面展开全面综述, 揭示这一抗噬菌体系统的微观奥秘。
1 CBASS系统概述
科学家们发现微生物抗噬菌体系统(如CRISPR-Cas系统和限制修饰系统)常常聚集在原核生物基因组的“防御岛”上, 并利用这一特性陆续发现了多个新型防御系统[11-14]。CBASS系统的发现与cGAS-STING通路的研究密切相关。cGAS蛋白作为通路的核心元件—细胞质中病毒DNA的感受器, 能够识别病毒DNA并生成cGAMP。cGAMP作为信号分子随后结合效应蛋白STING, 并诱导其发生寡聚化, 进而诱导I型干扰素和NF-κB信号通路, 从而引发保护性抗病毒免疫反应[15-19]。已有研究发现,在霍乱弧菌中也存在cGAMP, 它由霍乱弧菌二核苷酸环化酶(dinucleotide cyclase in Vibrio, DncV)合成,可以激活磷脂酶CapV, 促使其降解细菌内膜, 破坏膜的完整性, 最终导致细菌细胞死亡[20-24]。2019年,SOREK研究团队[9]注意到霍乱弧菌中编码DncV的基因同系物经常倾向于出现在防御基因附近, 于是通过对38 167个微生物基因组进行同源性搜索, 揭示了cGAMP信号是细菌中普遍存在的抗噬菌体防御系统的组成部分, 且该系统的多样化变体存在于超过10%的细菌与古细菌中。他们将该系统命名为CBASS。
生物信息学与功能分析研究表明, CBASS系统至少包含两种关键蛋白: 一是寡核苷酸环化酶(cGAS/DncV-like nucleotidyltransferase, CD-NTase),其核心功能是感知噬菌体的存在并产生环状寡核苷酸信号; 二是效应蛋白, 作为环寡核苷酸信号的受体, 被激活后会启动细胞杀伤功能。除了核心组分外, 还存在辅助蛋白, 有助于调节CD-NTase或激活效应蛋白。2020年, SOREK团队[25]对38 000类细菌和古细菌基因组进行分析, 鉴定出超过5 000种CBASS系统, 并根据操纵子构成将其分为4种主要类型(图1)。
I型CBASS。原核生物中最常见的构型, 结构相对简单, 仅由CD-NTase基因和效应蛋白基因组成,不含其他辅助基因。
II型CBASS。在CD-NTase和效应蛋白的基础上,额外包含Cap2和Cap3两个基因, 这两个基因编码的蛋白具有真核经典泛素化相关的结构域[26]。其中,Cap2含有E1和E2酶结构域, Cap3编码的结构域常见于真核生物的去泛素化酶中。
III型CBASS。除了基础的CD-NTase和效应蛋白外, 还包含编码HORMA和TRIP13结构域的辅助基因, 而这些结构域此前主要在真核生物中被发现[27]。该类型在变形菌门中大量存在, 而在厚壁菌门中几乎不存在, 显示出明显的物种分布差异。
IV型CBASS。主要集中在古细菌中, 除了基础基因外, 还包括具有核苷酸修饰结构域的辅助蛋白基因。例如, Cap9具有QueC酶结构域[28-29], Cap10属于TGT酶[30], 以及编码N-糖基化酶/DNA裂解酶的Cap11[31]等。
尽管在CBASS系统的多种构型中, 许多调控组分的具体功能和分子机制尚未被完全阐明, 但这些不同组分已被证实是构成各类CBASS构型不可或缺的部分。对CBASS系统的深入研究, 不仅能帮助我们更全面地理解细菌与噬菌体之间复杂的相互作用, 还能为开发新型抗菌策略(如利用噬菌体精准杀菌)和分子生物学工具(如基于CBASS组件的基因编辑工具)提供重要的理论基础, 具有广阔的科研和应用前景。
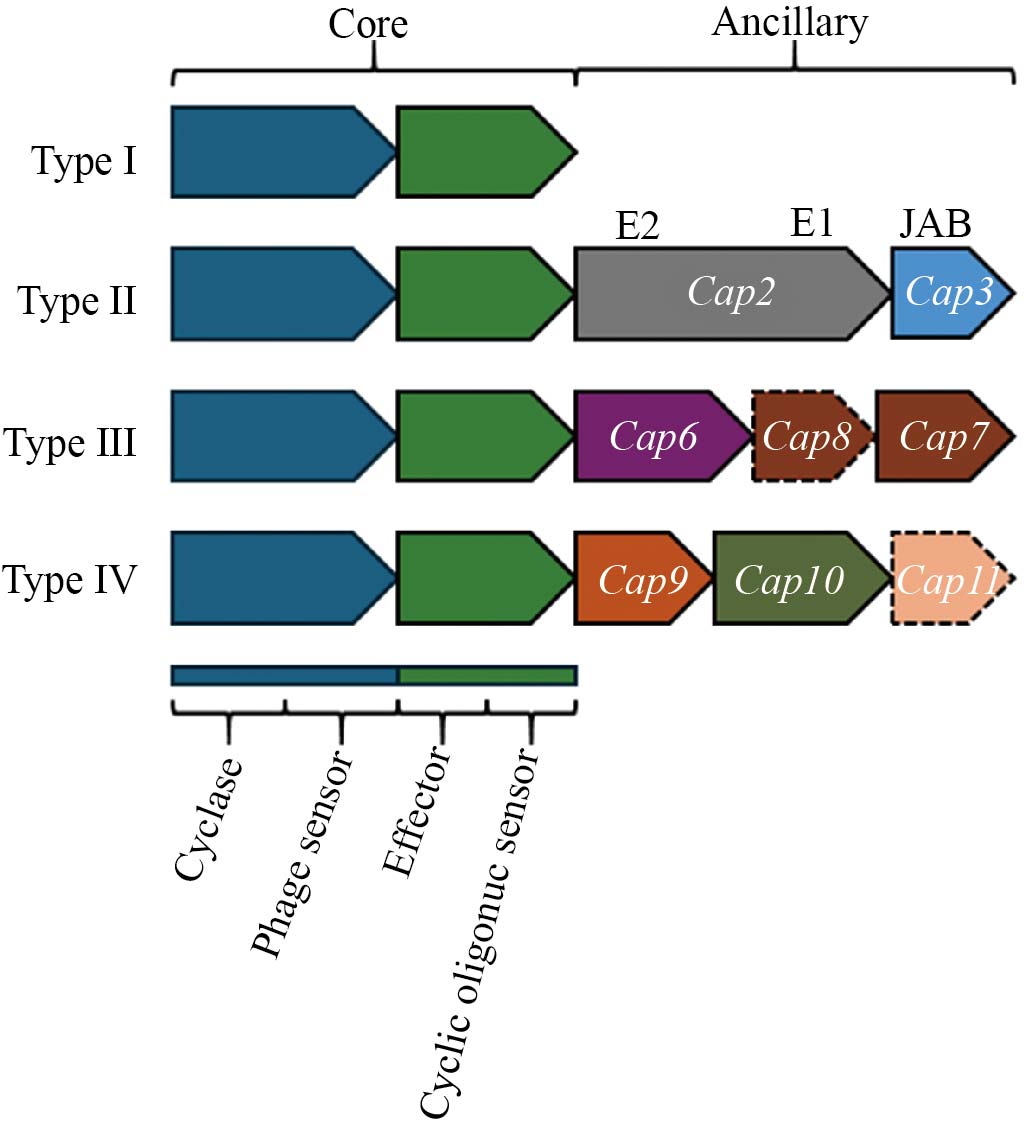
CBASS系统的核心部分包括寡核苷酸环化酶和效应蛋白。辅助基因被标记为Cap基因(虚线框表示该基因并不是存在该类型所有成员中)。
The core of the CBASS system consists of oligonucleotide cyclases and effector proteins. Ancillary genes are denoted as Cap genes (dashed boxes: genes variably absent in the type).
图1 CBASS系统分类(此图根据参考文献[25]改编)
Fig.1 Classification of the CBASS system (this figure is adapted from reference [25])
2 CBASS系统研究现状
2.1 环化酶的研究进展
CBASS环化酶被认为是cGAS的古老祖先, 但它们之间也有一些区别。在未感染的真核生物体内, DNA被限制在细胞核中, 只有在受到病毒攻击时, cGAS才能感知到细胞质中的病毒DNA并作出反应[15-19]。然而, 由于细菌没有细胞核, 如果CBASS仅仅对DNA的存在作出反应, 就势必会导致猖獗的自身免疫。CBASS环化酶究竟通过感知什么物质来启动下游免疫反应长期困扰着研究人员们。洛克菲勒大学的科研团队在相关研究中取得了令人瞩目的成果[32]。他们重点研究了施氏葡萄球菌(一种常见于狗、猫和其他动物口腔中的细菌, 在极少数情况下会传染给人类)中的CBASS系统, 发现葡萄球菌噬菌体能够产生一种特殊的cabRNA, 这种RNA分子具有独特的功能, 它能够结合CdnE03环化酶, 进而促进cGAMP的合成, 激活CBASS免疫反应[33]。这一发现揭示了噬菌体与细菌之间一种全新的相互作用模式, 为深入理解噬菌体感染过程以及细菌的免疫防御机制提供了新的视角。
细菌抗噬菌体系统通常对宿主细胞具有毒性,环化酶必须受到严格控制以防止激活不必要的细胞反应。环化酶的活性受到群体感应、细菌的代谢状态以及伴侣蛋白多种因素的动态调控。在霍乱弧菌中, 叶酸代谢中间产物二氢叶酸可通过结合DncV酶C-端的变构口袋, 诱导催化结构域的α5螺旋发生12°旋转, 从而封闭底物进入通道, 而细菌可通过群体感应和叶酸耗竭激活CBASS抗噬菌体系统[34-35]。CORBETT团队[36]表明在III型CBASS系统中, CBASS编码的HORMA结构域蛋白质在结合某些特定肽后由开放态转变为闭合态, 然后结合并激活CD-NTase以产生环状三腺苷酸第二信使, 进而激活核酸内切酶效应蛋白NucC, 导致细胞死亡和流产性感染; 而TRIP13的同源物可通过结合并分解活性HORMACD-NTase复合物, 抑制信号转导, 起到负调控的作用。2024年有研究发现在超过5 000个预测的CBASS中, 有2 199个CBASS编码泛素系统的同源物, 可分为两大类: 一是E1E2/JAB-CBASS, 由1 583个系统组成, 含有E1-E2融合蛋白(Cap2)和JAB去泛素化肽酶(Cap3); 第二种是E2-CBASS, 包含616个系统, 是一类短型CBASS, 仅含有E2蛋白[37]。E2-CBASS通过模仿泛素化级联来激活cGAS产生cGAMP, cGAMP作为抗病毒信号并导致细胞死亡[37]。同年, 研究表明Cap2蛋白可以将环化酶专一地连接到噬菌体休克蛋白(PspA)来抑制环化酶的活性, 当噬菌体侵染细菌后, 环化酶-PspA的结合可以被Cap3逆转, 从而释放环化酶, 并启动环化酶的活化[38]。
随着对cGAS-STING通路的深入研究, 环二核苷酸(cyclic dinucleotides, CDNs)类似物已成为免疫调节和癌症免疫治疗中具有前景的先导化合物[39-40]。CDNs最早是在细菌中被发现的, 然而人们对于细菌信号核苷酸产物所引发的免疫反应的真实范围的理解, 长期局限于环状二嘌呤分子。2019年, WHITELEY等[20]通过对细菌信号核苷酸进行系统筛选, 发现了一大类CD-NTases, 它们可以利用嘌呤和嘧啶核苷酸来合成各种类型的环二核苷酸(如cGAMP、cUAMP、cUUMP等)。通过系统性的晶体结构解析,研究确立了CD-NTases作为一个结构保守的蛋白家族, 并鉴定了其活性中心介导嘌呤/嘧啶底物选择性的关键氨基酸残基。有趣的是CD-NTase的产物不仅限于环二核苷酸, 还包括一类环状三核苷酸化合物(如cAAG、cAAA等)。
细菌CBASS系统与后生动物cGAS-STING通路共享核心机制, 提示环状寡核苷酸介导的免疫防御起源于细菌, 在后生动物中适应性进化。3’2’-cGAMP是目前发现的唯一同时存在于细菌和后生动物中的环状寡核苷酸, 在细菌中参与抗噬菌体防御, 在后生动物中激活抗病毒免疫, 体现功能进化保留性。而在后生动物中2’3’-cGAMP激活免疫能力更强, 体现后生动物免疫信号调控的适应性进化CBASS系统的环状寡核苷酸信号分子多样, 且与后生动物在进化上存在密切关联, 为深入理解生物免疫机制提供了重要视角。
2.2 CBASS系统效应蛋白研究现状
当噬菌体侵染细菌时, CD-NTase被激活生成环状寡核苷酸。效应蛋白的感应结构域识别这些环状寡核苷酸后, 效应蛋白随之被激活, 其细胞杀伤结构域开始发挥作用, 触发细胞的自杀性死亡, 从而抑制噬菌体的增殖(图2)。4种类型CBASS操纵子共享6种效应蛋白[25]。
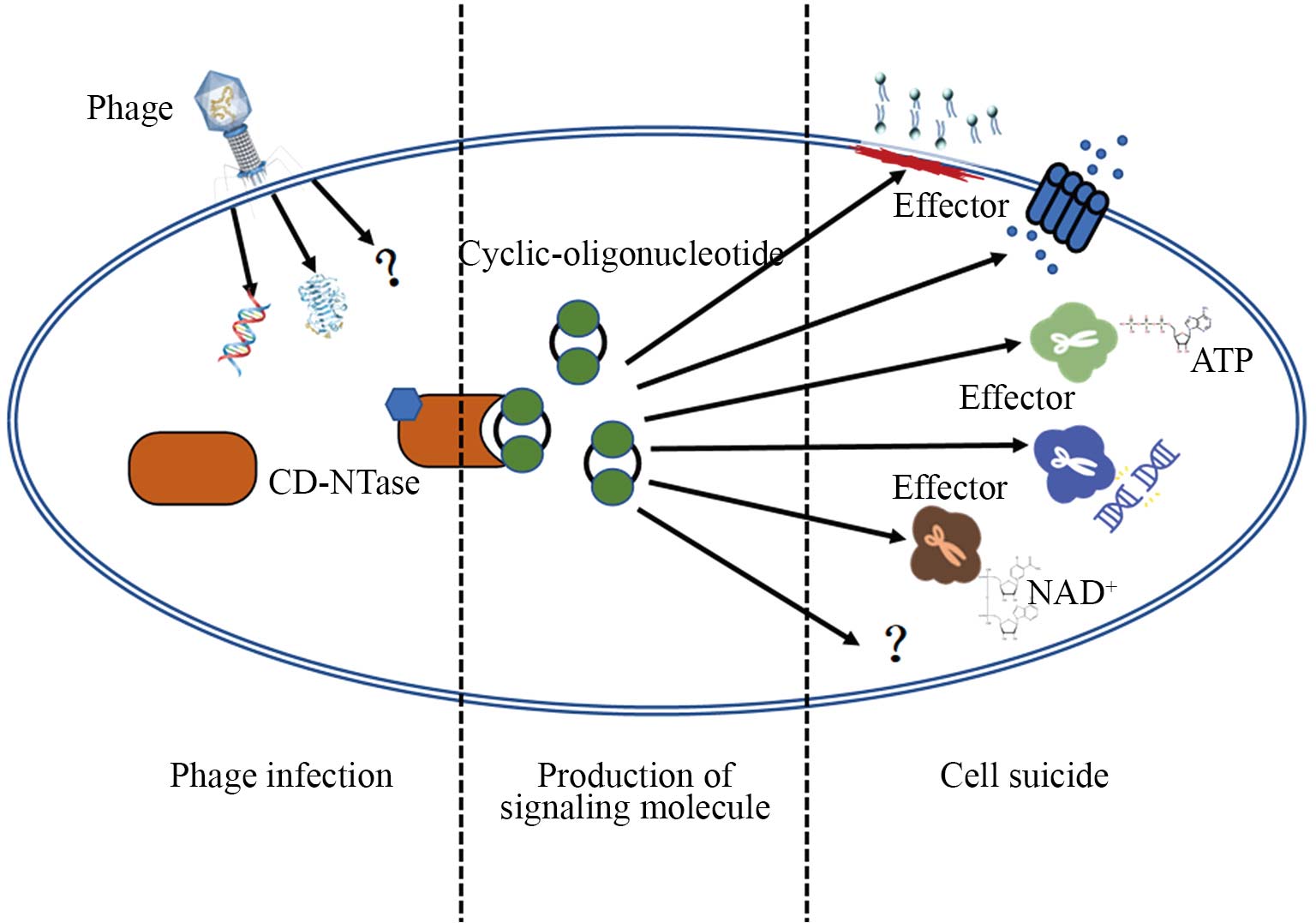
ATP: 腺嘌呤核苷三磷酸; NAD+: 烟酰胺腺嘌呤二核苷酸。
ATP: adenosine-triphosphate; NAD+: nicotinamide adenine dinucleotide.
图2 CBASS系统示意图
Fig.2 Model of CBASS system
2.2.1 具有跨膜螺旋(TM)的效应蛋白 具有2个跨膜螺旋(2TM)的效应蛋白广泛分布于I型和IV型CBASS系统中, 而具有4个跨膜螺旋(4TM)的效应蛋白则只在I、II、III型CBASS系统中有少量存在, 不存在于IV型CBASS系统中, 且可能是两个2TM效应蛋白的融合。2021年, KRANZUSCH团队[41]基于先前的生物信息学分析结果研究发现超过40%的CBASS操纵子编码TM效应蛋白, 这种效应形式的出现频率显著高于其他效应蛋白。他们以Cap15为主要研究对象, 该效应蛋白包含2个跨膜结构域, 且C-端融合了一个富含β折且功能未知的结构域, 晶体结构解析发现Cap15形成一个紧凑的8股β桶状结构, 仅在3’3’-cGAMP核苷酸第二信使信号存在时才会被激活并发生寡聚化, 然后在整个细胞内重新定位, 并特异性地诱导内膜破裂。此外, 在所有TM效应蛋白中, 唯一具有潜在酶活性结构域的是Cap16蛋白(包含一个NUDIX水解酶结构域), 但是活性位点(E154A)的突变并未影响其诱导细胞毒性的能力, 这进一步证实了不同家族CBASS跨膜效应蛋白通过破坏内膜完整性引发细胞死亡是一种普遍存在的防御机制。
2.2.2 核酸内切酶 这类效应蛋白表现出4种不同的内切核酸酶结构域亚类, 包括DUF4297核酸内切酶(Cap4)、NucC核酸内切酶、HNH核酸内切酶(Cap5)和钙调磷酸酶样核酸酶(Mrr)。除Mrr外, 其余3类核酸内切酶结构域效应蛋白均已被证实激活后可非特异性地将DNA降解成短片段, 导致噬菌体侵染失败并促进细胞死亡。2020年, KRANZUSCH团队[26]发现了Cap4的结构显示其通过配体诱导寡聚化激活DNA核酸内切酶, CBASS免疫使用与CARF相关的效应器感知3’-5’-和2’-5’-连接的环状寡核苷酸信号, 并保护细菌免受噬菌体感染。寡核苷酸识别是通过附加的SAVED结构域完成的, 该结构域是III型CRISPR免疫中两个CRISPR关联CARF亚基的意外融合。像锁和钥匙一样, SAVED效应器可精确区分2’-5’-和3’-5’-连接的细菌环状寡核苷酸信号, 并能够特异性识别至少180个潜在的核苷酸第二信使。效应蛋白NucC是同源三聚体的DNA核酸内切酶, 在被感染的细胞中, NucC会在三重对称的变构口袋中结合第二信使cAAA, 三聚体组装成同型六聚体,NucC被激活最终导致宿主细胞的死亡[24]。2021年,研究表明激活Cap5的HNH核酸酶结构域, 实现DNA降解, 依赖于3’2’-cGAMP介导的双SAVED结构域的“前后堆叠”(back-to-front stacking)[42]。
2.2.3 含有TIR结构域的效应蛋白 这类效应蛋白在4种类型的CBASS系统中存在均较少。TIR结构域最初被发现于真核生物中的先天免疫蛋白, 在CBASS系统中, TIR结构域通常与其他结构域融合发挥功能[43-45], 例如在I型CBASS系统中融合STING结构域, 以及在II型和III型CBASS系统中与SAVED结构域融合。这类效应蛋白依赖于融合结构域介导形成纤维状超分子复合物, 使相邻的TIR亚基相互作用, 组织和完成一个复合活性位点, 从而实现NAD⁺的降解。
2.2.4 具有patatin样磷脂酶结构域的效应蛋白 磷脂酶类效应蛋白在CBASS系统中发现最早, 广泛存在于I型和II型CBASS系统中, 其如何被激活并发挥细胞杀伤功能的分子机制尚不清楚, 也是领域内长期悬而未决的问题。我们近期的研究发现, 在未被激活的静息状态下, 来源于大肠杆菌的CBASS磷脂酶类效应蛋白CapE以非常紧密的二聚体形式存在,且通往酶反应中心的底物通道处于封闭状态[46]。当受到噬菌体感染时, 寡核苷酸环化酶CdnE被激活,利用ATP和UTP合成环二核苷酸信号分子cUMPAMP(简称cUA)。我们发现, CapE与cUA的亲和力极高(KD≈8.7 nmol/L)。当我们向CapE溶液中加入cUA后, 蛋白会从溶液状态转变为凝胶态。冷冻电镜结构分析表明, cUA的结合触发CapE发生关键构象变化, 使原本柔性的lid loop区发生显著构象变化,使结合的cUA被完全包裹在闭合口袋中。这一构象变化暴露出若干重要的位点, 使CapE以二聚体为组装单元, 以“头尾相接”的方式线性堆叠, 自组装形成纤维状超分子复合物。如多米诺骨牌效应一样, 纤维状复合物的组装诱导原本覆盖在底物通道入口处的α7螺旋发生构象改变, 使通往酶活中心的底物通道完全开放, 原本隐藏的催化位点暴露, 促使效应蛋白CapE从静息态转变为活化状态。活化后的CapE磷脂酶类效应蛋白能够高效裂解细胞膜, 最终导致细胞裂解和死亡。这些级联反应最终引发流产感染效应, 从而抑制噬菌体增殖(图3)。
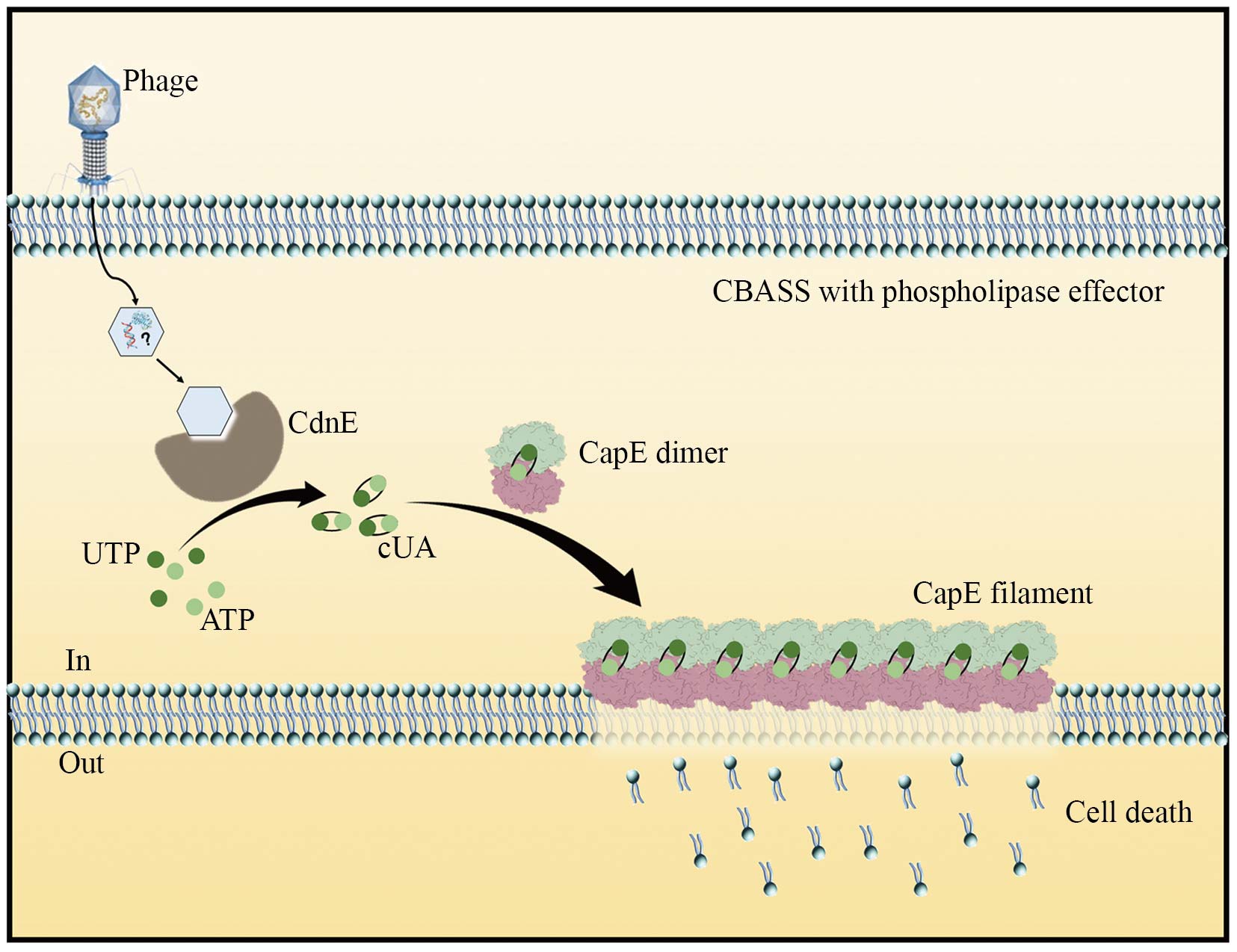
环二核苷酸结合并诱导无活性的CBASS磷脂酶效应物同源二聚体形成有活性的纤维状结构, 从而导致细胞膜被破坏, 进而阻止噬菌体传播。
The cyclic dinucleotide binds and induces the inactive homodimer of CBASS phospholipase effector to form an active filamentous structure, leading to membrane disruption and subsequent blockade of phage dissemination.
图3 CBASS磷脂酶类效应蛋白的抗噬菌体免疫机制(根据参考文献[46]改编)
Fig.3 The anti-phage immune mechanism of phospholipase-like effectors in CBASS (adapted from reference [46])
我们的研究通过对上述实验结果的分析, 确定了CapE在识别cUA、分子自组装及催化活化过程中的关键位点。随后, 结合定点突变及噬菌体感染实验, 系统地验证了这些关键位点对CapE抗噬菌体功能的重要性。为进一步模拟CapE被cUA激活后裂解细胞膜的过程, 我们采用脂质体模拟细胞膜结构, 并加入CapE与cUA进行电镜观察, 结果表明CapE在响应cUA后能够通过自组装介导膜裂解。此外, 我们还利用冷冻电子断层扫描(cryo-ET)和超分辨荧光成像, 观察到在噬菌体感染过程中, CapE在细菌细胞内聚集并形成纤维状结构的现象。
值得注意的是, 我们发现来源于霍乱弧菌的类似效应蛋白CapV(CapE同源蛋白)在结合第二信使环分子3’3’-cGAMP后, 同样可形成纤维状结构。这就提示通过超分子组装执行细胞杀伤功能可能是CBASS磷脂酶类效应蛋白的保守机制。更有趣的是,磷脂酶类效应蛋白在结合环状寡核苷酸后形成的超分子纤维结构, 与真核细胞中cGAS-STING通路激活后的多聚化组装现象非常相似。这就像是数亿年前的一次基因“分家”, 细菌选择直接裂解细胞阻断病毒传播, 而人类细胞则演化出更精细的免疫应答—但它们的核心逻辑惊人一致。
2.2.5 其他 CBASS系统效应蛋白还包括含有磷酸化酶/核苷酶超家族以及含有肽酶结构域两类, 它们分别在III型和I型CBASS系统中少量存在。目前关于这两类效应蛋白的研究还较少, 2023年,SOREK课题组[47]发现CBASS免疫效应蛋白Cap17是一种ATP核苷酸酶, 可在噬菌体感染期间裂解N-糖苷键以降解必需分子ATP和dATP以终止感染。
2.3 噬菌体的反击: 抑制CBASS系统的策略
在漫长的进化过程中, 细菌演化出多种多样的噬菌体防御系统, 而噬菌体也通过突变、抗性基因或抑制蛋白等策略, 不断突破宿主防御, 形成复杂的“军备竞赛”。
2022年, 来自美国哈佛医学院的研究人员通过对大肠杆菌的多种噬菌体进行生化筛选, 发现来自T4噬菌体的Acb1蛋白可以通过广泛识别和降解环状二核苷酸和环状三核苷酸等核苷酸“第二信使”来阻断下游信号通路, 进而阻止被噬菌体感染的宿主菌的自杀, 逃逸宿主菌CBASS免疫来突破细菌防御, 其对3’腺苷残基的水解机制已由Acb1与3’3’-cGAMP复合物的晶体结构表明[48]。
2023年有研究团队发现铜绿假单胞菌含有cGAS样酶CdnA, 其可产生3’3’-cGAMP, 激活CapV,发挥CBASS系统的免疫功能, 响应PaMx41噬菌体感染; 而PaMx41噬菌体的逃逸突变体可产生一种抗CBASS系统的蛋白Acb2[49]。他们发现Acb2可与3’3’-cGAMP分子具有超高亲和力, 但不具有酶活,并不能像Acb1那样通过降解环状核苷酸来阻断下游信号通路。结构显示Acb2与3’3’-cGAMP分子通过稳定的相互作用实现紧密结合—Acb2蛋白自身形成紧凑的六聚体结构, 每两个Acb2单体N-端形成“结合口袋”包裹住一个3’3’-cGAMP分子。Acb2通过紧密结合3’3’-cGAMP分子, 阻断其激活CapV,有效地抑制了CBASS系统的免疫作用。后续通过体外生化实验, 他们又进一步证实了Acb2还可以结合c-di-AMP、3’3’-c-di-UMP、2’3’-cGAMP等多种寡核苷酸信号分子, 覆盖I型和II型两种CBASS系统,显示出Acb2是一种基于cGAS酶的免疫系统的广谱抑制蛋白。
3 讨论与展望
噬菌体的侵袭威胁催生了细菌多样化的防御策略, 其中CBASS系统尤为关键。该系统通过合成环化核苷酸第二信使(如cGAMP)激活下游效应蛋白, 形成广谱抗病毒防御网络。CBASS系统中的环化酶(CD-NTase)属于cGAS/DncV样核苷酸转移酶家族, 其SAVED效应器通过锁和钥匙的原理精确区分不同连接的寡核苷酸信号, 该机制与CRISPR-Cas免疫系统中的CARF结构域存在进化保守性[26,32]。更值得注意的是, CBASS系统与高等生物的cGASSTING通路存在明确的进化同源性, 暗示这些免疫机制可能起源于原核生物并随生命演化分化。例如,细菌CBASS系统中的磷脂酶类效应蛋在结合环状寡核苷后发生构象变化, 并自组装形成超分子纤维状结构, 从而开放底物通道, 暴露催化位点, 高效裂解细胞膜以阻断噬菌体复制, 与真核细胞中cGASSTING通路激活后的多聚化现象高度相似, 体现了数亿年前基因“分家”后, 细菌选择通过直接裂解自身细胞阻止病毒传播, 而人类细胞则演化出更精细的干扰素介导的免疫应答, 但两者的核心逻辑高度一致—通过信号分子触发防御机制[50-51]。但是,细菌通过CBASS环化酶可以感知噬菌体感染过程中产生的特异性RNA结构, 而非像真核生物的cGAS系统那样依赖病毒DNA的错误定位。
尽管CBASS系统的防御潜力显著, 其研究仍面临诸多未解之谜。一方面, 其核心环化酶(CD-NTase)在体外表现出组成性活性, 但如何在体内被精确激活以避免自身免疫, 如何调控产生不同连接方式的环化核苷酸以精准激活下游效应蛋白, 以及如何通过移植CBASS系统实现跨细菌的抗噬菌体防御, 仍是领域内长期悬而未决的问题。另一方面, 不同类型的效应蛋白是如何执行其杀伤细菌或抑制其生长的功能也尚未被完全阐明。同时, CBASS系统的研究对于未来噬菌体疗法的实现以及作为分子生物学工具研发库的研究都有巨大潜力, 应用前景广阔, 例如通过工程化改造其核心元件, 可开发噬菌体感染的实时检测系统; 或是用于噬菌体感染的早期防控及新型细胞破碎工具的开发。然而, 这些应用依赖于对其激活机制、功能转移以及结构域协同作用的全面解析, 未来研究有望进一步揭示CBASS系统的复杂性, 为抗病毒策略和分子工具设计提供理论依据。
参考文献 (References)
[1] BIKARD D, MARRAFFINI L A. Innate and adaptive immunity in bacteria: mechanisms of programmed genetic variation to fight bacteriophages [J]. Curr Opin Immunol, 2012, 24(1): 15-20.
[2] LABRIE S J, SAMSON J E, MOINEAU S. Bacteriophage resistance mechanisms [J]. Nat Rev Microbiol, 2010, 8(5): 317-27.
[3] KOONIN E V, MAKAROVA K S, WOLF Y I. Evolutionary genomics of defense systems in archaea and bacteria [J]. Annu Rev Microbiol, 2017, 71: 233-61.
[4] ROSTØL J T, MARRAFFINI L. (Ph)ighting phages: how bacteria resist their parasites [J]. Cell Host Microbe, 2019, 25(2): 184-94.
[5] VAN HOUTE S, BUCKLING A, WESTRA E R. Evolutionary ecology of prokaryotic immune mechanisms [J]. Microbiol Mol Biol Rev, 2016, 80(3): 745-63.
[6] DY R L, RICHTER C, SALMOND G P C, et al. Remarkable mechanisms in microbes to resist phage infections [J]. Annu Rev Virol, 2014, 1(1): 307-31.
[7] VAN DER OOST J, WESTRA E R, JACKSON R N, et al. Unravelling the structural and mechanistic basis of CRISPR-Cas systems [J]. Nat Rev Microbiol, 2014, 12(7): 479-92.
[8] WESTRA E R, SWARTS D C, STAALS R H J, et al. The CRISPRs, they are a-changin’: how prokaryotes generate adaptive immunity [J]. Annu Rev Genet, 2012, 46: 311-39.
[9] COHEN D, MELAMED S, MILLMAN A, et al. Cyclic GMPAMP signalling protects bacteria against viral infection [J]. Nature, 2019, 574(7780): 691-5.
[10] ZAVER S A, WOODWARD J J. Cyclic dinucleotides at the forefront of innate immunity [J]. Curr Opin Cell Biol, 2020, 63: 49-56.
[11] MAKAROVA K S, WOLF Y I, SNIR S, et al. Defense islands in bacterial and archaeal genomes and prediction of novel defense systems [J]. J Bacteriol, 2011, 193(21): 6039-56.
[12] GOLDFARB T, SBERRO H, WEINSTOCK E, et al. BREX is a novel phage resistance system widespread in microbial genomes [J]. EMBO J, 2015, 34(2): 169-83.
[13] OFIR G, MELAMED S, SBERRO H, et al. DISARM is a widespread bacterial defence system with broad anti-phage activities [J]. Nat Microbiol, 2018, 3(1): 90-8.
[14] DORON S, MELAMED S, OFIR G, et al. Systematic discovery of antiphage defense systems in the microbial pangenome [J]. Science, 2018, 359(6379): eaar4120.
[15] KRANZUSCH P J, WILSON S C, LEE A S Y, et al. Ancient origin of cGAS-STING reveals mechanism of universal 2’,3’cGAMP signaling [J]. Mol Cell, 2015, 59(6): 891-903.
[16] MARGOLIS S R, WILSON S C, VANCE R E. Evolutionary origins of cGAS-STING signaling [J]. Trends Immunol, 2017, 38(10): 733-43.
[17] SUN L, WU J, DU F, et al. Cyclic GMP-AMP synthase is a cytosolic DNA sensor that activates the type I interferon pathway [J]. Science, 2013, 339(6121): 786-91.
[18] ABLASSER A, GOLDECK M, CAVLAR T, et al. cGAS produces a 2’-5’-linked cyclic dinucleotide second messenger that activates STING [J]. Nature, 2013, 498(7454): 380-4.
[19] ISHIKAWA H, MA Z, BARBER G N. STING regulates intracellular DNA-mediated, type I interferon-dependent innate immunity [J]. Nature, 2009, 461(7265): 788-92.
[20] WHITELEY A T, EAGLESHAM J B, DE OLIVEIRA MANN C C, et al. Bacterial cGAS-like enzymes synthesize diverse nucleotide signals [J]. Nature, 2019, 567(7747): 194-9.
[21] DAVIES B W, BOGARD R W, YOUNG T S, et al. Coordinated regulation of accessory genetic elements produces cyclic dinucleotides for V. cholerae virulence [J]. Cell, 2012, 149(2): 358-70.
[22] SEVERIN G B, RAMLIDEN M S, HAWVER L A, et al. Direct activation of a phospholipase by cyclic GMP-AMP in El Tor Vibrio cholerae [J]. Proc Natl Acad Sci USA, 2018, 115(26): E6048-E55.
[23] BURROUGHS A M, ZHANG D, SCHÄFFER D E, et al. Comparative genomic analyses reveal a vast, novel network of nucleotide-centric systems in biological conflicts, immunity and signaling [J]. Nucleic Acids Res, 2015, 43(22): 10633-54.
[24] LAU R K, YE Q, BIRKHOLZ E A, et al. Structure and mechanism of a cyclic trinucleotide-activated bacterial endonuclease mediating bacteriophage immunity [J]. Molecular Cell, 2020, 77(4): 723-33,e6.
[25] MILLMAN A, MELAMED S, AMITAI G, et al. Diversity and classification of cyclic-oligonucleotide-based anti-phage signalling systems [J]. Nat Microbiol, 2020, 5(12): 1608-15.
[26] LOWEY B, WHITELEY A T, KESZEI A F A, et al. CBASS immunity uses CARF-related effectors to sense 3’-5’- and 2’-5’-linked cyclic oligonucleotide signals and protect bacteria from phage infection [J]. Cell, 2020, 182(1): 38-49,e17.
[27] VADER G. Pch2(TRIP13): controlling cell division through regulation of HORMA domains [J]. Chromosoma, 2015, 124(3): 333-9.
[28] READER J S, METZGAR D, SCHIMMEL P, et al. Identification of four genes necessary for biosynthesis of the modified nucleoside queuosine [J]. J Biol Chem, 2004, 279(8): 6280-5.
[29] MCCARTY R M, SOMOGYI A, LIN G, et al. The deazapurine biosynthetic pathway revealed: in vitro enzymatic synthesis of PreQ(0) from guanosine 5’-triphosphate in four steps [J]. Biochemistry, 2009, 48(18): 3847-52.
[30] OKADA N, NOGUCHI S, KASAI H, et al. Novel mechanism of post-transcriptional modification of tRNA. Insertion of bases of Q precursors into tRNA by a specific tRNA transglycosylase reaction [J]. J Biol Chem, 1979, 254(8): 3067-73.
[31] DAVID S S, O’SHEA V L, KUNDU S. Base-excision repair of oxidative DNA damage [J]. Nature, 2007, 447(7147): 941-50.
[32] BANH D V, ROBERTS C G, MORALES-AMADOR A, et al. Bacterial cGAS senses a viral RNA to initiate immunity [J]. Nature, 2023, 623(7989): 1001-8.
[33] MISIC A M, CAIN C L, MORRIS D O, et al. Complete genome sequence and methylome of staphylococcus schleiferi, an important cause of skin and ear infections in veterinary medicine [J]. Genome Announc, 2015, 3(5): e01011-15.
[34] ZHU D, WANG L, SHANG G, et al. Structural biochemistry of a Vibrio cholerae dinucleotide cyclase reveals cyclase activity regulation by folates [J]. Molecular Cell, 2014, 55(6): 931-7.
[35] SEVERIN G B, RAMLIDEN M S, FORD K C, et al. Activation of a Vibrio cholerae CBASS anti-phage system by quorum sensing and folate depletion [J]. mBio, 2023, 14(5): e0087523.
[36] YE Q, LAU R K, MATHEWS I T, et al. HORMA domain proteins and a Trip13-like ATPase regulate bacterial cGAS-like enzymes to mediate bacteriophage immunity [J]. Mol Cell, 2020, 77(4): 709-22,e7.
[37] YAN Y, XIAO J, HUANG F, et al. Phage defence system CBASS is regulated by a prokaryotic E2 enzyme that imitates the ubiquitin pathway [J]. Nat Microbiol, 2024, 9(6): 1566-78.
[38] KRÜGER L, GASKELL-MEW L, GRAHAM S, et al. Reversible conjugation of a CBASS nucleotide cyclase regulates bacterial immune response to phage infection [J]. Nat Microbiol, 2024, 9(6): 1579-92.
[39] CORRALES L, GLICKMAN L H, MCWHIRTER S M, et al. Direct activation of STING in the tumor microenvironment leads to potent and systemic tumor regression and immunity [J]. Cell Rep, 2015, 11(7): 1018-30.
[40] FU J, KANNE D B, LEONG M, et al. STING agonist formulated cancer vaccines can cure established tumors resistant to PD-1 blockade [J]. Sci Transl Med, 2015, 7(283): 283ra52.
[41] DUNCAN-LOWEY B, MCNAMARA-BORDEWICK N K, TAL N, et al. Effector-mediated membrane disruption controls cell death in CBASS antiphage defense [J]. Mol Cell, 2021, 81(24): 5039-51,e5.
[42] FATMA S, CHAKRAVARTI A, ZENG X, et al. Molecular mechanisms of the CdnG-Cap5 antiphage defense system employing 3’,2’-cGAMP as the second messenger [J]. Nat Commun, 2021, 12(1): 6381.
[43] MOREHOUSE B R, GOVANDE A A, MILLMAN A, et al. STING cyclic dinucleotide sensing originated in bacteria [J]. Nature, 2020, 586(7829): 429-33.
[44] HOGREL G, GUILD A, GRAHAM S, et al. Cyclic nucleotideinduced helical structure activates a TIR immune effector [J]. Nature, 2022, 608(7924): 808-12.
[45] MOREHOUSE B R, YIP M C J, KESZEI A F A, et al. Cryo-EM structure of an active bacterial TIR-STING filament complex [J]. Nature, 2022, 608(7924): 803-7.
[46] WANG J, LI Z, LANG H, et al. Cyclic-dinucleotide-induced filamentous assembly of phospholipases governs broad CBASS immunity [J]. Cell, 2025, 188(14): 3744-56,e16.
[47] ROUSSET F, YIRMIYA E, NESHER S, et al. A conserved family of immune effectors cleaves cellular ATP upon viral infection [J]. Cell, 2023, 186(17): 3619-31,e13.
[48] HOBBS S J, WEIN T, LU A, et al. Phage anti-CBASS and antipycsar nucleases subvert bacterial immunity [J]. Nature, 2022, 605(7910): 522-6.
[49] HUITING E, CAO X, REN J, et al. Bacteriophages inhibit and evade cGAS-like immune function in bacteria [J]. Cell, 2023, 186(4): 864-76,e21.
[50] PATEL D J, YU Y, XIE W. cGAMP-activated cGAS-STING signaling: its bacterial origins and evolutionary adaptation by metazoans [J]. Nat Struct Mol Biol, 2023, 30(3): 245-60.
[51] BURDETTE D L, VANCE R E. STING and the innate immune response to nucleic acids in the cytosol [J]. Nat Immunol, 2013, 14(1): 19-26.



 中文版
中文版 英文版
英文版