领域前沿·中国
ProTracer—不间断记录体内细胞增殖的遗传示踪新技术
石梦杨 何灵娟 周斌*
( 中国科学院分子细胞科学卓越创新中心/生物化学与细胞生物学研究所, 中国科学院大学,上海 200031)
| 周斌, 中国科学院分子细胞科学卓越创新中心研究员。2002年毕业于浙江大学医学院临床医学系并获得硕士学位; 2006年毕业于中国医学科学院中国协和医科大学并获得博士学位; 2006年至2010年在美国哈佛大学医学院波士顿儿童医院从事博士后研究; 2010年9月起至2016年8月任中国科学院上海生命科学研究院营养科学研究所研究员; 2016年9月起至今任职于中国科学院生物化学与细胞生物学研究所研究员。周斌研究组主要开发遗传谱系示踪新技术, 研究哺乳动物体内细胞起源及命运调控机制, 代表性研究工作多次在Nature、Science、Nat Med、Nat Genet、Circulation、Cell Stem Cell、Dev Cell等国际学术期刊上发表。其中发现“哺乳动物新生期心脏具有重新生成冠状动脉的能力”入选2014年度“中国科学十大进展”; 创建的谱系示踪新技术解决了近20年来国际心血管领域内关于心脏干细胞与心肌再生的争议问题; 开发了细胞增殖的遗传学示踪技术, 从全新的角度揭示了器官修复再生的调控机制。周斌研究员曾获得国际心脏研究会ISHR“杰出研究员奖”、“谈家桢生命科学创新奖”、科学探索奖等多项奖励。 |
ProTracer—不间断记录体内细胞增殖的遗传示踪新技术
石梦杨 何灵娟 周斌*
(细胞生物学国家重点实验室, 中国科学院分子细胞科学卓越创新中心,
中国科学院上海生物化学与细胞生物学研究所, 中国科学院大学, 上海 200031)
[摘要] 器官内细胞数量和组织稳态可以通过细胞增殖调节并维持。大多数用于鉴别具有显著增殖能力的细胞的相关研究, 都是基于某一细胞亚群的谱系示踪, 而这会导致潜在的选择性偏差。该团队利用双同源重组酶系统, 开发了一种示踪增殖细胞的新技术—ProTracer, 该技术能够实现在多个器官中长时间不间断地记录整个细胞类群的细胞增殖。在肝脏中, ProTracer揭示了位于肝小叶中间区域的肝细胞在生理稳态、损伤修复和再生过程中具有优越的增殖能力。克隆分析实验结果表明, 大部分ProTracer标记的肝细胞发生了细胞分裂。综上, ProTracer技术通过遗传记录总细胞群的增殖事件, 能够应用于无偏好性检测具有显著再生能力的独特细胞亚群。
[关键词] 细胞增殖; 谱系示踪; ProTracer; 双同源重组酶系统; 器官; 肝脏; 遗传记录; 再生
[Abstract] Organ homeostasis is orchestrated by timed and spatially restricted cell proliferation. Studies identifying cells with superior proliferative capacity mostly rely on lineage tracing of a subset of cell populations, introducing a potential selective bias. Here, this group developed a genetic system (proliferation tracer, ProTracer) by incorporating dual recombinases to seamlessly record proliferation events of entire cell populations over time in multiple organs. In the liver, ProTracer revealed that a subpopulation of midzonal hepatocytes have superior proliferative capacity during homeostasis, injury repair, and regeneration. Clonal analysis showed that the majority of hepatocytes labeled by ProTracer have undergone cell division. By genetically recording proliferation events of entire cell populations, ProTracer enables unbiased detection of unique cellular compartments with enhanced regenerative capacity.
[Keywords] proliferation; lineage tracing; ProTracer; dual recombinases; organs; liver; genetically recording; regeneration
1 肝细胞再生研究背景
体内器官稳态往往是由高度增殖的细胞来维持的[1-4]。这种特殊细胞大多有特定的标记基因, 并且得到了谱系示踪技术的证实[5-8]。然而, 使用单个标记基因只能定义某一种细胞类群, 从而会导致实验分析更聚焦于带有该标记的细胞命运, 而不会上升至总体细胞群水平上, 因而会存在潜在偏差。故由此而来的不同遗传命运图谱会得到相互矛盾的结论, 正如当前一些关于肝细胞再生来源的具有高度争议的研究[9-18]。有研究表明, 门静脉周围的肝细胞能够逐渐向中央静脉迁移[9]。然而, 这个从门静脉向中央静脉方向的肝细胞流动模型仍存在争议[10-11]。有谱系示踪研究表明, 位于肝小叶中央静脉周围的Axin2+肝细胞, 会从中央静脉至门静脉方向扩增, 维持肝脏稳态时的肝细胞更新[12]。然而, 其他谱系示踪研究团队却发现, 中央静脉周围的Axin2+或Lgr5+肝细胞对肝脏稳态维持和再生的贡献有限[17-18]。也有研究团队发现, 在肝损伤后, 肝门静脉周围的Sox9+肝细胞广泛增殖并修复受损肝脏[13]。此外, 有一些研究表明, 新生肝细胞的来源并非集中于某个区域, 而是分散在整个区域, 比如有研究报道分布在肝小叶各个区域的LGR4+肝细胞具有较强的扩增能力维持肝脏稳态[14], 另有研究报道, 在肝小叶所有分区中都有表达的端粒酶逆转录酶(telomerase reverse transcriptase, TERT)高表达的肝细胞比TERT低表达的肝细胞具有更强的增殖能力[16], 这些支持了有关肝细胞再生的一种“分布式模型”。最近有关肝细胞扩增的克隆分析表明, 在处于稳态和再生过程的肝脏中, 肝细胞之间的增殖能力无明显差异[19]。在稳态和再生过程中, 肝细胞的更新主要是通过已有肝细胞的增殖来维持的[20], 而在某些严重损伤的情况下, 肝细胞可以被胆管细胞的转化所取代[21-24]。鉴于肝小叶不同区域的肝细胞增殖能力具有异质性, 故当前的关键问题应该围绕不同肝细胞亚群对肝脏稳态和再生的贡献展开, 由于上述依赖单个标记基因或稀疏标记的谱系示踪研究追踪的只是肝细胞某个亚群, 不同的标记得出不同的结果, 使得目前很难判断新生肝细胞的真正来源。因此, 长时间不间断地对整个肝细胞类群的增殖进行的无偏好性检测, 可以阐明在肝脏稳态和损伤修复过程中新生肝细胞的来源。
2 细胞增殖遗传记录系统的设计与构建
Ki67是一种广泛使用的细胞增殖标记物[25], 近期报道的两种基于Ki67-CreER的谱系示踪方法是在注射他莫昔芬(Tamoxifen, Tam)后诱导标记Ki67+增殖细胞[25-26]。若要有效维持小鼠体内Cre酶活性, 需要进行连续几周或几个月的Tam注射, 但这可能会对组织产生毒性和副作用[27]。为此, 我们开发了一种基于双同源重组酶系统的策略, 实现通过单次Tam注射能够长时间不间断地记录细胞增殖事件。该遗传系统的构建涉及3种小鼠品系: DreER[28]、Ki67-CrexER(Ki67-Cre-rox-ER-rox)和R26-GFP[29](图1)。
我们首先构建并鉴定了Ki67-CrexER小鼠品系。Ki67和ESR(代表CrexER的表达)共染结果证实, CrexER的表达准确反映Ki67的表达。此外, 核酸类似物EdU掺入实验表明, CrexER和EdU的染色结果相似, 表明Ki67-CrexER表达在增殖细胞里。接着, 体外培养并监测Ki67-CrexER;R26-GFP小鼠细胞的增殖情况。 CrexER与CreER一样, 可以通过Tam注射后, 经由Cre-loxP重组发挥作用, 不同的是CrexER能通过Dre-rox重组转换为Cre[30]。活体成像结果显示, Ki67-CrexER;R26-GFP小鼠细胞经Tam诱导后, 增殖细胞表达GFP, 且单个表达GFP的细胞正经历细胞分裂。Ki67-CrexER;R26-GFP小鼠主动脉三维全器官成像显示, GFP+内皮细胞几乎总是成对出现。上述实验结果表明, Ki67-CrexER;R26-GFP标记的是增殖的细胞。此外, 我们发现, Ki67-CrexER/CrexER纯合小鼠正常生长, 且细胞正常增殖, 提示Ki67-CrexER等位基因维持Ki67内源性表达。
该系统的精妙之处在于, 通过单剂量注射Tam启动该系统后, 它将永远保持打开状态, 从而可以在此后的任何时间窗口内如实且特异地记录细胞增殖情况。我们将该系统模型称为ProTracer(proliferation tracer)。
3 ProTracer可以有效记录体内细胞增殖
ProTracer小鼠(R26-DreER[28];Ki67-CrexER; R26-GFP)能够在大多数器官和组织中遗传记录增殖细胞。通过Southern blot对ProTracer小鼠的多个组织的基因组DNA分析, 我们发现Tam诱导后, DreER将Ki67-CrexER转变为Ki67-Cre效率为50%~100%, 且肝脏中最高。在经由Tam注射激活系统后的第2天和第28天, 全器官荧光成像和GFP免疫荧光检测结果表明, GFP信号仅来自发生Cre-loxP重组的Ki67+细胞。
为便于将ProTracer系统与先前报道的细胞增殖示踪技术进行比较, 我们还构建出Ki67-CreER;R26-GFP小鼠。经Tam注射后的第2天, ProTracer和Ki67-CreER;R26-GFP小鼠所有被检测组织(除了高增殖的肠上皮)GFP信号均很少。而Tam实验后第28天, ProTracer小鼠的GFP信号明显多于Ki67-CreER;R26-GFP小鼠。这一结果说明了ProTracer长时间持续捕捉细胞增殖事件的能力。
在没有Tam处理的情况下, Ki67-CreER;R26- GFP和ProTracer小鼠中几乎检测不到任何GFP+细胞。且Tam处理的R26-DreER;R26-GFP小鼠中也没有任何GFP+细胞, 排除了系统中潜在的DreER-loxP重组。这些研究结果表明, Tam处理的ProTracer小鼠不同器官或组织中检测到的GFP+细胞依赖于DreER-rox重组和随后的Cre-loxP重组。综上, 这些结果突出了ProTracer能够实现长时间不间断地记录不同组织器官中的细胞增殖。
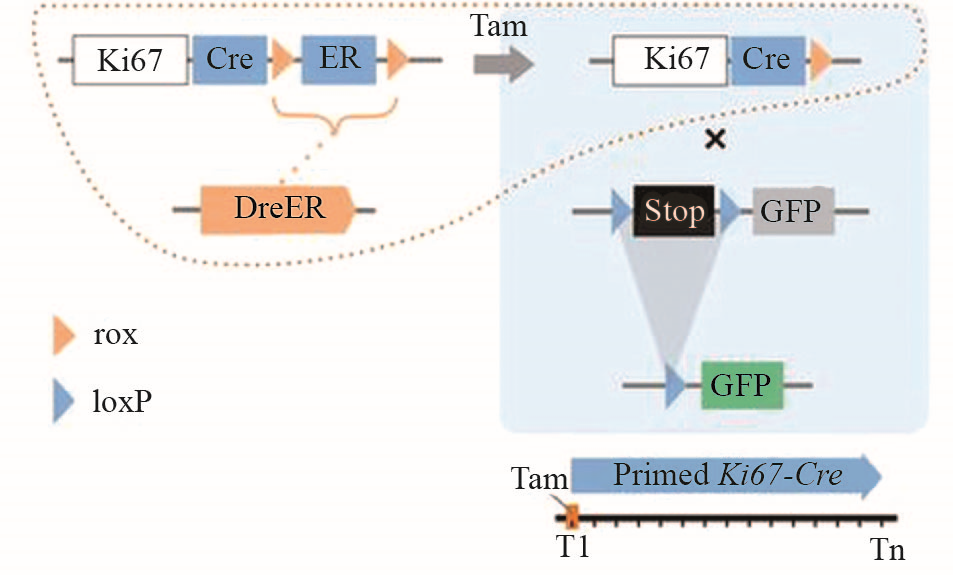
Tam诱导的DreER-rox重组将Ki67启动子驱动的CrexER转换为Cre, 启动系统持续记录从T1到Tn间所有的细胞增殖事件。
Tam-induced DreER-rox recombination switches CrexER into Cre driven by the Ki67 promoter, which primes the system to continuously record all cell proliferation events from T1 to Tn.
图1 ProTracer策略示意图(根据参考文献[31]修改)
Fig.1 Schematic showing the ProTracer strategy (modified from reference [31])
4 ProTracer系统对生理稳态情况下肝细胞增殖能力鉴定
我们利用ProTracer系统来检测所有肝细胞群的增殖, 研究新生肝细胞产生的来源和位置。肝小叶被分为三个不同的区域[32-34]: 门静脉周围区[分子标记为E-钙黏蛋白(E-cadherin, E-CAD)]为1区; 最靠近中央静脉的中央区域(由谷氨酰胺合成酶GS标记)为3区; 其余的中间区域(E-CAD–GS–)为2区[35](图2A)。Southern blot结果表明, 经Tam注射后, ProTracer小鼠肝细胞中几乎所有Ki67-CrexER都转换为了Ki67-Cre, 而溶剂玉米油处理的对照组没有。我们还通过将R26-DreER与rox报告小鼠R26-rox-Stop-rox-tdTomato (R26-RSR-tdTomato)杂交, 发现经Tam注射后R26-DreER重组发生在肝脏的几乎所有肝细胞中。
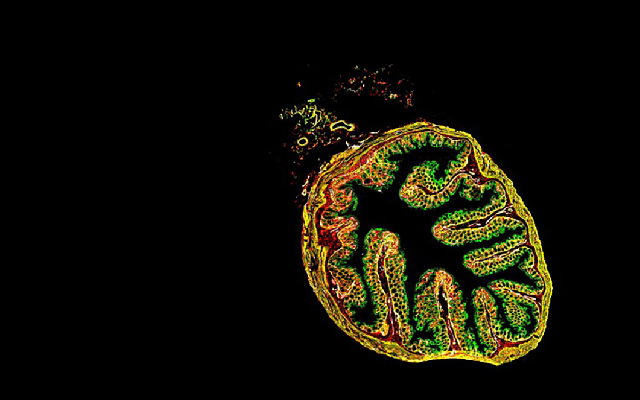
为检测肝小叶原位肝细胞增殖情况, 我们在Tam诱导后不同时间点采集ProTracer小鼠的肝脏样本。第0天和第2天免疫荧光染色结果显示, 肝切片上没有或鲜有GFP+肝细胞。从第2周到第8周, 观察到逐渐增多的GFP+肝细胞, 且主要集中在2区。在第8~12周, 我们检测到1区GFP+肝细胞数量增加, 但其密度仍显著低于2区。肝小叶中2区GFP+肝细胞明显增多, 提示该区域有更多新的肝细胞生成。值得注意的是, 在第4~12周, 3区GFP+细胞的数量没有显著变化(图2B)。于是, 我们进一步使用GS-CreER驱动系统独立检测在肝脏稳态期间, 3区肝细胞是否显著扩张。通过使用GS-flagBFP和GS-CreER敲入小鼠系, 我们证实了GS特异性表达于中央周围肝细胞即3区肝细胞。然而, 对这些GS+肝细胞的遗传示踪结果显示, 它们在8周之后并没有显著扩大或减少。谱系示踪和ProTracer数据均表明, 3区肝细胞在肝脏稳态期间不会显著扩张。综上所述, 这些基于对总肝细胞群监测的数据表明, 在肝脏稳态期间, 肝细胞增殖率在2区最高, 1区中等, 3区最低。
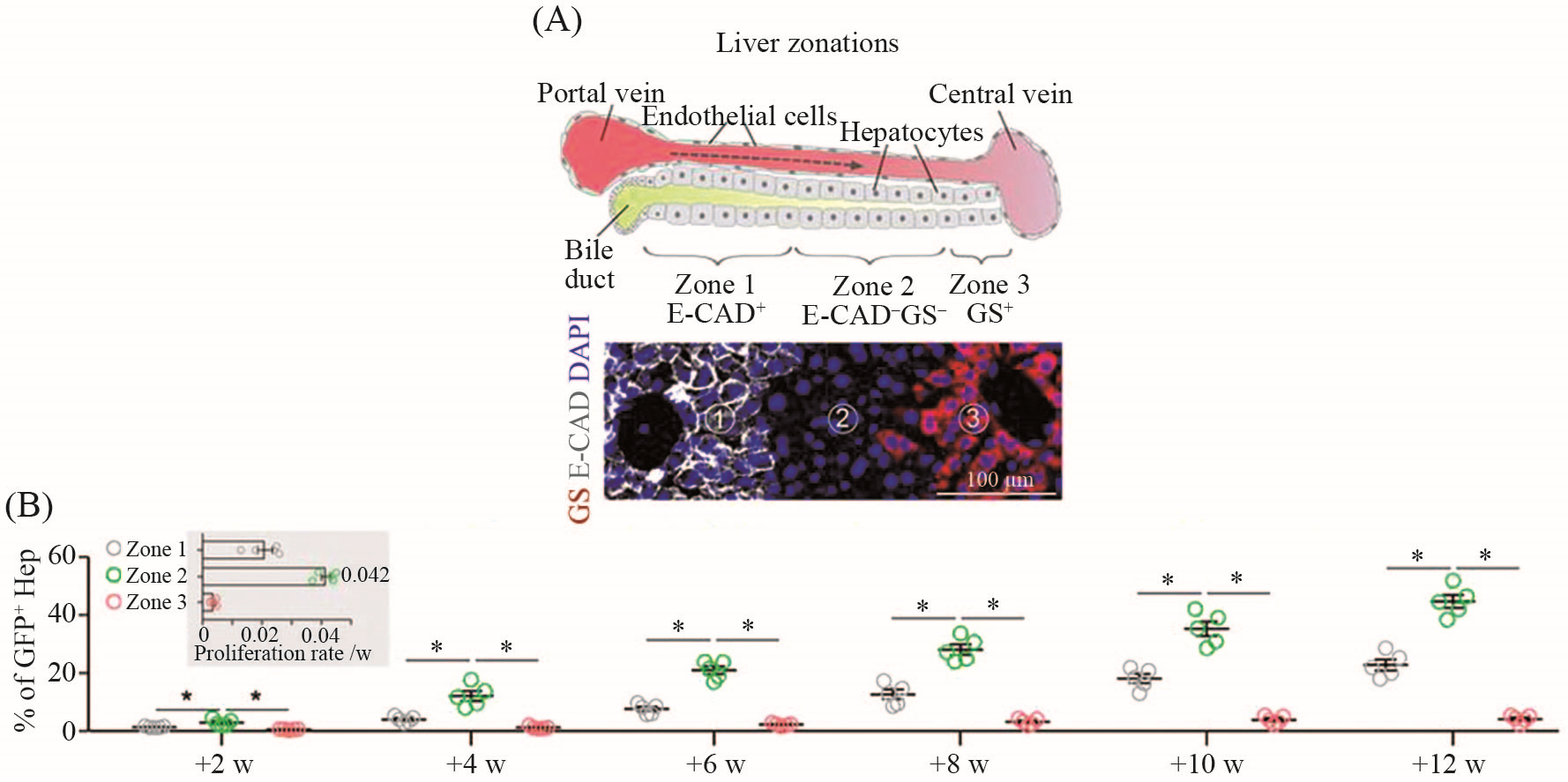
A: 上图从门静脉周围到中央区的三个肝分区。下图为谷氨酰胺合成酶(GS)和钙黏蛋白(E-CAD)免疫荧光染色。①、②、③分别表示1区(E-CAD+)、2区(E-CAD–GS–)和3区(GS+)。虚线箭头表示血流方向。B: 肝小叶各区域表达绿色荧光蛋白的肝细胞(Hep)百分比。数据为x_±s; n=5。*P<0.05。插图为每个区域5个时间窗(第2周至第4周、第4周至第6周、第6周至第8周、第8周至第10周和第10周至第12周)中肝细胞的增殖率。每个数值代表五个单独的生物样本。
A: three liver zones from the periportal to the pericentral regions. Lower panel shows immunostaining for GS (glutamine synthetase) and E-CAD (E-cadherin). ①, ② and ③ indicate zone 1 (E-CAD+), zone 2 (E-CAD–GS–), and zone 3 (GS+), respectively. Dashed arrow indicates blood flow. B: quantification of the percentage of Hep (hepatocytes) expressing GFP in each zone of the liver lobule. Data are x_±s; n=5. *P<0.05. Inset is proliferation (proli.) rate of hepatocytes per week from five time windows (weeks 2 to 4, 4 to 6, 6 to 8, 8 to 10, and 10 to 12) in each zone. Each figure is representative of five individual biological samples.
图2 肝脏的三个分区及增殖情况(根据参考文献[31]修改)
Fig.2 Three liver zones and regional hepatocyte proliferation (modified from reference [31])
5 肝细胞特异性ProTracer揭示肝脏中明显的“甜甜圈”型增殖模式
为了更清楚地辨别整个器官细胞的增殖模式并特异地监测肝细胞谱系, 我们对ProTracer系统进行下一步优化, 将Alb-DreER[36]小鼠与含有双同源重组酶激活的报告基因Ai66的Ki67-CrexER[37]小鼠杂交(图3A)。经Tam注射后, Alb-DreER在肝脏的所有三个分区中均有效地重组肝细胞。Alb-DreER;Ki67-CrexER;Ai66小鼠肝脏切片免疫荧光染色分析结果证实, 所有tdTomato+细胞为Fah+和HNF4a+肝细胞, 而非CK19+导管细胞、Desmin+星状细胞、PDGFRa+成纤维细胞、VE-CAD+内皮细胞和F4/80+巨噬细胞。以上结果证明了由肝细胞特异性ProTracer记录的是特异性肝细胞的增殖, 而不是其他类型细胞的增殖。
我们在Tam诱导Alb-DreER;Ki67-CrexER;Ai66小鼠后的第6周收集了小鼠肝脏样品。肝脏全器官荧光成像结果显示, 肝细胞特异性的tdTomato+增殖信号以独特的类似甜甜圈形状呈现(图3B)。用tdTomato、GS和E-CAD抗体对肝脏切片进行免疫荧光染色, 证实了这种高度区域性的增殖结果, 并表明了大多数具有增殖活性的肝细胞发生在2区(图3C和图3D)。作为技术对照, 在没有注射Tam的情况下, Alb-DreER;Ki67-CrexER;Ai66小鼠肝脏中未观察到任何tdTomato+肝细胞。
在已知AAV8-TBG-Dre(AAV8-Dre)具有强的肝细胞趋向性[38]的基础上, 为避免经Tam注射引起的潜在偏差, 我们通过给Ki67-CrexER;R26-GFP小鼠注射AAV8-Dre来在肝细胞中特异性地激活ProTracer系统。在AAV注射8周后的小鼠肝脏中, GFP+细胞呈肝细胞特异性标志物HNF4a+和FAH+。肝脏切片免疫荧光染色结果表明, 大部分GFP+肝细胞位于2区, 而1区或3区明显较少, 进一步证实了在肝脏稳态期间观察到的中间区域性肝细胞增殖的结果。
我们还探讨了在长期研究过程(如几周到几个月)中, ProTracer系统是否可以扩展为活体小鼠中肝细胞增殖事件的无创监测。我们用荧光素酶小鼠替代荧光报告基因小鼠, 并使用AAV8-Dre[38]实现肝细胞特异性ProTracer激活。在AAV8-Dre诱导的ProTracer激活后, 对同一只活小鼠每两周进行荧光素酶信号监测, 我们发现AAV8-Dre组的荧光素酶活性逐渐增加, 而AAV8-对照组没有。这表明随着时间的推移, 肝细胞增殖逐渐增加。该数据还进一步表明, 肝脏中增殖的甜甜圈样信号是在组织稳态过程中肝细胞增殖逐渐积累的结果。
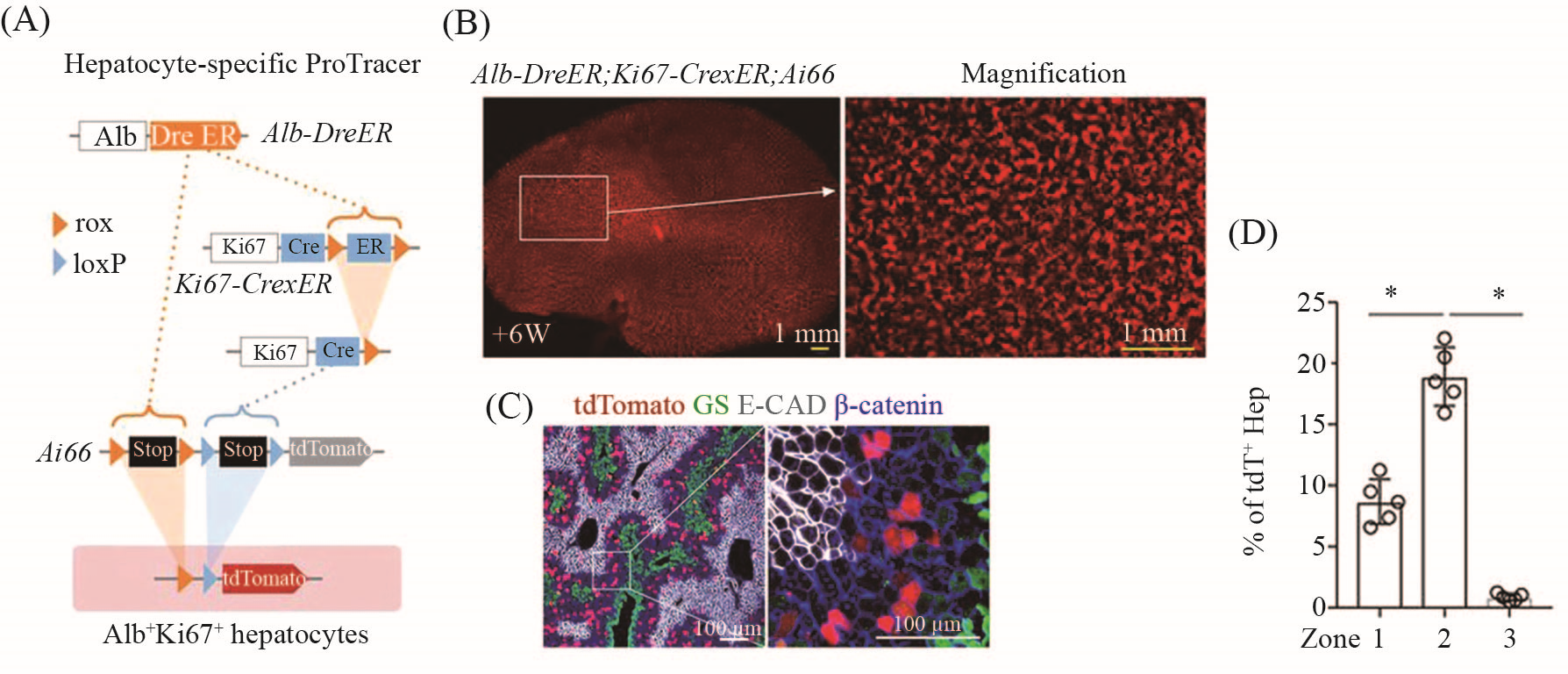
A: 肝细胞特异性ProTracer系统的实验策略。B: Tam诱导肝脏特异性ProTracer小鼠6周后, 肝脏全器官荧光成像结果图。C: 对肝切片进行tdTomato、GS、E-CAD和β-catenin免疫荧光染色图。D: tdTomato+肝细胞在肝小叶各区域的百分比。数据为x_±s; n= 5。*P<0.05。
A: the experimental strategy used for hepatocyte-specific tracing of cell proliferation. B: whole-mount fluorescence image of liver collected from hepatocyte-specific ProTracer mouse at six weeks after Tam induction. C: immunostaining for tdTomato, GS, E-CAD, and β-catenin on liver sections. D: quantification of the percentage of hepatocytes expressing tdTomato in each zone of the liver lobule. Data are x_±s; n=5. *P<0.05.
图3 肝细胞特异性ProTracer系统(根据参考文献[31]修改)
Fig.3 Hepatocyte-specific ProTracer (modified from reference [31])
6 大多数被ProTracer标记的Ki67+肝细胞发生了细胞分裂
大多数肝细胞是多倍体和双核的[39], 因此, 表达细胞周期标记物(如Ki67)并不一定代表肝细胞发生了细胞分裂, 有可能是发生了多倍体化或者核分裂[40-41]。为确定ProTracer标记的Ki67+细胞进行细胞分裂和多倍体化的比例, 我们用低剂量AAV8-Dre对Ki67-CrexER;R26-GFP小鼠中Ki67+肝细胞进行稀疏GFP标记。通过稀疏标记实验, 我们发现存在成对的邻近的GFP+肝细胞, 表明这些细胞发生了细胞分裂, 而单个的GFP+肝细胞有2个或2个以上细胞核表明是多倍体而没有细胞分裂。为了在三维肝小叶中准确检测这些事件, 我们在50~100 μm厚的肝切片上分析了多个免疫荧光染色Z轴叠图, 结果表明大多数GFP+细胞显示为一对相邻的肝细胞。连续切片和定量数据分析显示, 超过90%的GFP+肝细胞呈2个及以上的细胞聚集状态, 单独GFP+的肝细胞的占比最小。同样, 这些肝细胞大部分位于肝小叶的2区。以上数据表明, ProTracer标记的Ki67+肝细胞大部分发生了细胞分裂。
7 基于Ccna2的ProTracer显示肝细胞高度区域性增殖
为证实上述观察结果, 我们利用曾用于检测细胞增殖的第二个细胞周期基因Ccna2[42]构建并培育Ccna2-CrexER小鼠。我们将Tam注射到R26-DreER; Ccna2-CrexER;R26-GFP小鼠体内, 6周后采集肝脏进行分析。肝切片免疫荧光染色结果显示, GFP信号在2区更为明显。定量结果显示, 与1区和3区相比, 2区肝细胞增殖率明显更高。
为了进一步增加独立于Tam但能特异性标记肝细胞Ccna2的ProTracer分析, 我们用AAV-Dre病毒诱导Ccna2-CrexER;R26-GFP小鼠, 并在12周后收集肝脏。全器官荧光成像显示出饱满的GFP+信号的圆形图案。免疫荧光染色肝脏切片及定量数据结果显示, GFP+肝细胞密度在2区最高, 3区最低, 这一结果与我们基于Ki67的ProTracer实验一致。综上所述, 基于ProTracer的细胞增殖基因(Ki67和Ccna2)记录显示了肝细胞在肝稳态期间增殖的空间异质性, 并揭示了肝小叶中肝细胞增殖的高度区域性。
8 肝损伤和再生情况下肝细胞增殖情况
成人肝细胞在受伤的情况下发生自我增殖, 以替代损坏的肝细胞维持细胞稳态, 因而具有显著的再生能力[3]。为研究肝再生过程中肝细胞的增殖, 我们首先进行了部分肝切除术(partial hepatectomy, PHx)模型, 这是一种不引入分区偏差的损伤模型[43]。在Tam诱导ProTracer小鼠1周后进行PHx手术, 在手术后的第24、40天小鼠和2、3、4、7天分析肝脏样本。PHx后第1天至第7天, 全器官荧光成像图显示肝脏中的GFP+信号逐渐增强。免疫荧光染色发现PHx后的第24 h鲜有GFP+肝细胞。PHx后的第40 h, GFP+的肝细胞首先出现在1区, PHx后的第2天, GFP+肝细胞平缓增加, 大部分位于1区和2区。PHx后的第3天和第4天, 肝小叶中GFP+肝细胞显著增加, 大部分位于2和1区, 2区肝细胞增殖已高于1区。第7天, 所有肝区均检测到GFP+肝细胞, 大多数肝细胞标记在1区和2区, 3区部分肝细胞未被标记(图4A)。
为记录3区在肝损伤后修复和再生过程中的增殖事件, 我们对ProTracer小鼠使用导致3区肝细胞急性损伤的四氯化碳(CCl4)处理。在CCl4处理后的第2、4、7天, 我们收集ProTracer小鼠的肝脏样本进行分析。全器官荧光成像显示, CCl4处理后第2天至第7天, 肝脏中GFP+信号增加。肝切片的免疫荧光染色结果显示, CCl4处理后第2天和第4天, 2区富集了GFP+肝细胞。第7天, 2区和3区大多数肝细胞为GFP+。考虑到CCl4主要导致3区肝细胞死亡, 可能是2区肝细胞在第2天率先参与修复或再生过程, 并在第4天至第7天弥补3区肝细胞的损失。此外, 我们对ProTracer小鼠进行1区损伤模型胆管结扎手术(bile duct ligation, BDL), 并在BDL损伤后的第3、4、7天收集肝脏进行分析。我们发现, 在BDL模型中, 门静脉周围区损伤, GFP+肝细胞在2区高度富集, 诱导导管反应(图4B)。综上所述, 这些数据也表明, 损伤后肝脏修复和再生过程中均呈现肝细胞生成能力的高度区域性。
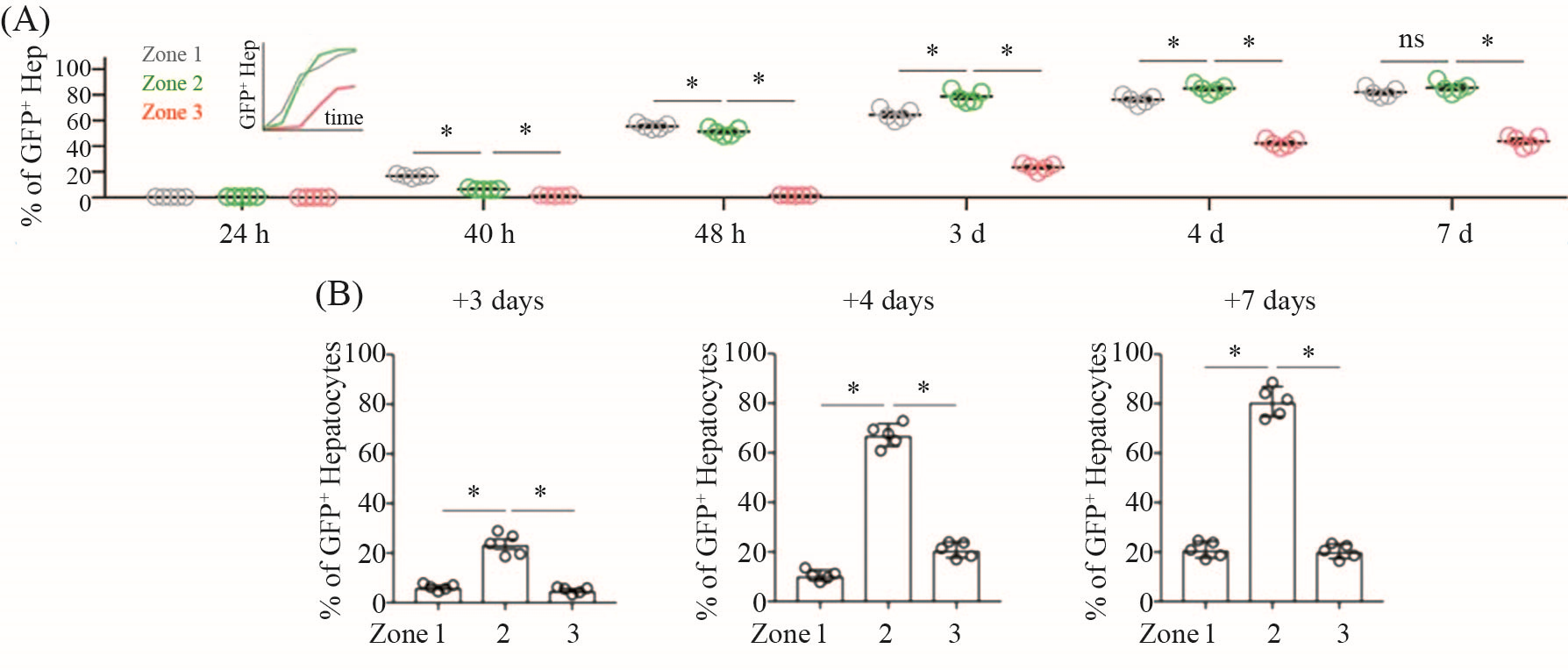
A、B分别为PHx手术模型和BDL手术模型下, 肝小叶各区域表达绿色荧光蛋白的肝细胞(Hep)百分比。数据为x_±s; n=5。*P<0.05。ns: 无统计学差异。A显示GFP+肝细胞百分比随PHx手术后时间推进的趋势。B显示的BDL手术后相关的数据。
Both A and B are quantification of the percentage of hepatocytes (Hep) expressing GFP in each zone of the liver lobule. Data are x_±s; n=5. *P<0.05; ns: nonsignificant. A indicates the trend of GFP+ hepatocyte percentage over time on the basis of quantification data after PHx. B indicates the data analysed after BDL.
图4 用ProTracer检测肝再生过程中的肝细胞增殖(根据参考文献[31]修改)
Fig.4 Hepatocyte proliferation examined by ProTracer during liver regeneration (modified from reference [31])
9 应用与展望
ProTracer系统能监测和记录在稳态和疾病背景下的组织细胞增殖事件。ProTracer策略有三个突出的特点值得强调。第一, 它允许对动物寿命中的任何时间窗口正在进行的细胞增殖事件进行长时间无缝隙记录(图2B和图4A), 从而实现数月至数年的长期研究。第二, 它可以用于研究某一特定细胞系的增殖。这对于研究极少增殖的细胞类型(如心肌细胞或神经元)尤其有益。与传统的基于化学和免疫荧光染色的方法相比, ProTracer能提高信噪比和分辨率, 将稀有的感兴趣细胞的增殖信号从其他增殖细胞背景中分离出来, 从而特异性的检测该细胞增殖。第三, ProTracer允许对活体动物细胞增殖进行无创的长期监测, 可以不间断地在几个星期、几个月甚至更长时间的窗口内检测同一动物的细胞增殖动态。
肝脏再生领域的一个关键问题是不同肝细胞亚群对肝脏稳态和再生的贡献。不同的研究得出的结论不同[12,17,44]。相较于这些依赖于单独标记基因的谱系示踪研究, ProTracer系统支持对给定类型的所有细胞进行群体水平分析。与最近的研究结果一致[17-18,44], 我们的实验数据不支持之前提出的Axin2+中心周肝细胞是肝干细胞的观点[12]。之前还有研究表明, 所有区域的肝细胞对肝脏稳态的贡献相同[14,16,18], 与此不同, ProTracer系统分析显示, 位于2区的肝细胞群在整个肝小叶中产生更多的新生肝细胞, 形成我们在各种增殖记录实验中观察到的类似于甜甜圈的形状。其他研究可能受限于不同肝细胞亚群的谱系示踪带来的潜在偏差或各自模型的低标记效率, 故没有识别出这个区域。而依赖于EdU掺入或Ki67染色的相关区域性肝细胞增殖分析的传统方法, 记录的时间可能太短, 以至于无法检测到我们基于ProTracer系统展开的研究中观察到的细胞增殖情况[14,18]。同样, 在稳态情况下, ProTracer系统的短期记录没有观察到肝细胞显示出区域增殖优势, 而需要连续记录数周至数月, 这可能是因为肝细胞在体内稳态时的低更新率[18]。ProTracer系统进一步表明, 在部分肝脏修复过程中, 2区肝细胞优先增殖, 这可能是因为大多数损伤发生在邻近的1区和3区[45]。而后续也有其他研究团队利用不同技术进一步证实, 肝内稳态由肝小叶中2区肝细胞维持[46]。
综上所述, 我们的发现以及ProTracer系统将使未来的研究能够进一步剖析不同肝细胞的再生潜力。而ProTracer系统的未来应用和进一步优化将极大地促进我们对细胞生成及其在多个器官发育、生长、再生和疾病中的动态变化的理解与研究。
参考文献 (References)
[1] MICHALOPOULOS G K, DEFRANCES M C. Liver re-generation [J]. Science, 1997, 276(5309): 60-6.
[2] BLANPAIN C, SIMONS B D. Unravelling stem cell dynamics by lineage tracing [J]. Nat Rev Mol Cell Biol, 2013, 14(8): 489-502.
[3] MIYAJIMA A, TANAKA M, ITOH T. Stem/progenitor cells in liver development, homeostasis, regeneration, and reprogramming [J]. Cell Stem Cell, 2014, 14(5): 561-74.
[4] MERRELL A J, STANGER B Z. Adult cell plasticity in vivo: de-differentiation and transdifferentiation are back in style [J]. Nat Rev Mol Cell Biol, 2016, 17(7): 413-25.
[5] CAI C L, MARTIN J C, SUN Y, et al. A myocardial lineage derives from Tbx18 epicardial cells [J]. Nature, 2008, 454(7200): 104-8.
[6] BECK B, DRIESSENS G, GOOSSENS S, et al. A vascular niche and a VEGF-Nrp1 loop regulate the initiation and stemness of skin tumours [J]. Nature, 2011, 478(7369): 399-403.
[7] VAN KEYMEULEN A, ROCHA A S, OUSSET M, et al. Distinct stem cells contribute to mammary gland development and maintenance [J]. Nature, 2011, 479(7372): 189-93.
[8] LARSIMONT J C, YOUSSEF K K, SANCHEZ-DANES A, et al. Sox9 controls self-renewal of oncogene targeted cells and links tumor initiation and invasion [J]. Cell Stem Cell, 2015, 17(1): 60-73.
[9] ZAJICEK G, OREN R, WEINREB M Jr. The streaming liver [J]. Liver, 1985, 5(6): 293-300.
[10] BRALET M P, BRANCHEREAU S, BRECHOT C, et al. Cell lineage study in the liver using retroviral mediated gene transfer. Evidence against the streaming of hepatocytes in normal liver [J]. Am J Pathol, 1994, 144(5): 896-905.
[11] ZAJICEK G. Do livers “stream” [J]? Am J Pathol, 1995, 146(3): 772-6.
[12] WANG B, ZHAO L, FISH M, et al. Self-renewing diploid Axin2+ cells fuel homeostatic renewal of the liver [J]. Nature, 2015, 524(7564): 180-5.
[13] FONT-BURGADA J, SHALAPOUR S, RAMASWAMY S, et al. Hybrid periportal hepatocytes regenerate the injured liver without giving rise to cancer [J]. Cell, 2015, 162(4): 766-79.
[14] PLANAS-PAZ L, ORSINI V, BOULTER L, et al. The RSPO-LGR4/5-ZNRF3/RNF43 module controls liver zonation and size [J]. Nat Cell Biol, 2016, 18(5): 467-79.
[15] PU W, ZHANG H, HUANG X, et al. Mfsd2a+ hepatocytes repopulate the liver during injury and regeneration [J]. Nat Commun, 2016, 7: 13369.
[16] LIN S, NASCIMENTO E M, GAJERA C R, et al. Distributed hepatocytes expressing telomerase repopulate the liver in homeostasis and injury [J]. Nature, 2018, 556(7700): 244-8.
[17] ANG C H, HSU S H, GUO F, et al. Lgr5+ pericentral hepatocytes are self-maintained in normal liver regeneration and susceptible to hepatocarcinogenesis [J]. Proc Natl Acad Sci USA, 2019, 116(39): 19530-40.
[18] SUN T, PIKIOLEK M, ORSINI V, et al. AXIN2+ pericentral hepatocytes have limited contributions to liver homeostasis and regeneration [J]. Cell Stem Cell, 2020, 26(1): 97-107,e6.
[19] CHEN Q, LIU Y, JEONG H W, et al. Apelin+ endothelial niche cells control hematopoiesis and mediate vascular regeneration after myeloablative injury [J]. Cell Stem Cell, 2019, 25(6): 768-83,e6.
[20] MATSUMOTO T, WAKEFIELD L, TARLOW B D, et al. In vivo lineage tracing of polyploid hepatocytes reveals extensive proliferation during liver regeneration [J]. Cell Stem Cell, 2020, 26(1): 34-47,e3.
[21] RAVEN A, LU W Y, MAN T Y, et al. Cholangiocytes act as facultative liver stem cells during impaired hepatocyte regeneration [J]. Nature, 2017, 547(7663): 350-4.
[22] DENG X, ZHANG X, LI W, et al. Chronic liver injury induces conversion of biliary epithelial cells into hepatocytes [J]. Cell Stem Cell, 2018, 23(1): 114-22,e3.
[23] SHIN S, UPADHYAY N, GREENBAUM LE, et al. Ablation of Foxl1-Cre-labeled hepatic progenitor cells and their descendants impairs recovery of mice from liver injury [J]. Gastroenterology, 2015, 148(1): 192-202,e3.
[24] RUSSELL J O, LU W Y, OKABE H, et al. Hepatocyte-specific beta-catenin deletion during severe liver injury provokes cholangiocytes to differentiate into hepatocytes [J]. Hepatology, 2019, 69(2): 742-59.
[25] KRETZSCHMAR K, POST Y, BANNIER-HELAOUET M, et al. Profiling proliferative cells and their progeny in damaged murine hearts [J]. Proc Natl Acad Sci USA, 2018, 115(52): E12245-54.
[26] BASAK O, KRIEGER T G, MURARO M J, et al. Troy+ brain stem cells cycle through quiescence and regulate their number by sensing niche occupancy [J]. Proc Natl Acad Sci USA, 2018, 115(4): E610-9.
[27] YANG G, NOWSHEEN S, AZIZ K, et al. Toxicity and adverse effects of Tamoxifen and other anti-estrogen drugs [J]. Pharmacol Ther, 2013, 139(3): 392-404.
[28] LI Y, HE L, HUANG X, et al. Genetic lineage tracing of nonmyocyte population by dual recombinases [J]. Circulation, 2018, 138(8): 793-805.
[29] ZHANG H, PU W, LIU Q, et al. Endocardium contributes to cardiac fat [J]. Circ Res, 2016, 118(2): 254-65.
[30] PU W, HE L, HAN X, et al. Genetic targeting of organ-specific blood vessels [J]. Circ Res, 2018, 123(1): 86-99.
[31] HE L, PU W, LIU X, et al. Proliferation tracing reveals regional hepatocyte generation in liver homeostasis and repair [J]. Science, 2021, 371(6532): eabc4346.
[32] HALPERN K B, SHENHAV R, MATCOVITCH-NATAN O, et al. Single-cell spatial reconstruction reveals global division of labour in the mammalian liver [J]. Nature, 2017, 542(7641): 352-6.
[33] JUNGERMANN K, KIETZMANN T. Zonation of parenchymal and nonparenchymal metabolism in liver [J]. Annu Rev Nutr, 1996, 16: 179-203.
[34] COLNOT S, PERRET C. Liver zonation [M]. US: Springer, 2011: 7-16.
[35] AIZARANI N, SAVIANO A, SAGAR, et al. A human liver cell atlas reveals heterogeneity and epithelial progenitors [J]. Nature, 2019, 572(7768): 199-204.
[36] WANG Y, HUANG X, HE L, et al. Genetic tracing of hepatocytes in liver homeostasis, injury, and regeneration [J]. J Biol Chem, 2017, 292(21): 8594-604.
[37] MADISEN L, GARNER A R, SHIMAOKA D, et al. Transgenic mice for intersectional targeting of neural sensors and effectors with high specificity and performance [J]. Neuron, 2015, 85(5): 942-58.
[38] YANGER K, KNIGIN D, ZONG Y, et al. Adult hepatocytes are generated by self-duplication rather than stem cell differentiation [J]. Cell Stem Cell, 2014, 15(3): 340-9.
[39] MARGALL-DUCOS G, CELTON-MORIZUR S, COUTON D, et al. Liver tetraploidization is controlled by a new process of incomplete cytokinesis [J]. J Cell Sci, 2007, 120(20): 3633-9.
[40] MIYAOKA Y, EBATO K, KATO H, et al. Hypertrophy and unconventional cell division of hepatocytes underlie liver re-generation [J]. Curr Biol, 2012, 22(13): 1166-75.
[41] KATSUDA T, HOSAKA K, MATSUZAKI J, et al. Transcriptomic dissection of hepatocyte heterogeneity: linking ploidy,zonation, and stem/progenitor cell characteristics [J]. Cell Mol Gastroenterol Hepatol, 2020, 9(1): 161-83.
[42] HIRAI M, CHEN J, EVANS S M. Tissue-specific cell cycle indicator reveals unexpected findings for cardiac myocyte proliferation [J]. Circ Res, 2016, 118(1): 20-8.
[43] MITCHELL C, WILLENBRING H. A reproducible and well-tolerated method for 2/3 partial hepatectomy in mice [J]. Nat Protoc, 2008, 3(7): 1167-70.
[44] CHEN F, JIMENEZ R J, SHARMA K, et al. Broad distribution of hepatocyte proliferation in liver homeostasis and regeneration [J]. Cell Stem Cell, 2020, 26(1): 27-33,e4.
[45] FORBES S J, NEWSOME P N. Liver regeneration-mechanisms and models to clinical application [J]. Nat Rev Gastroenterol Hepatol, 2016, 13(8): 473-85.
[46] WEI Y, WANG Y G, JIA Y, et al. Liver homeostasis is maintained by midlobular zone 2 hepatocytes [J]. Science, 2021, 371(6532): eabb1625.




 中文版
中文版 英文版
英文版